新品发布 || 新格元CeleLens单细胞数据分析模块
发布时间:2021-10-29 18:00:08
单细胞测序技术因其独有的高分辨率优势,吸引众多科研、临床和新药研究者的关注。自单细胞测序技术问世以来涌现出大量的单细胞分析方法和软件,并且持续快速地迭代。在单细胞测序数据维度高、分析难度大、可选方法参差不齐的现状下,如何选择最适合数据特征的分析方法?如何最小化单细胞数据分析对编程能力的要求?如何将数据以直观美观、描述性好的方式呈现,且无缝衔接科研杂志发表?解决这些问题是有效应用单细胞测序技术的先决条件。
自创始以来,新格元致力于提供优质的个性化分析服务为研究者解决以上问题,最大化单细胞测序的效益。为了进一步帮助研究者有效挖掘单细胞测序数据,新格元汇总2000+项目、1亿+细胞的个性化分析经验,实测60+种单细胞分析软件和主流算法,将成熟优质的单细胞数据分析管线打造成CeleLens标准化分析模块,缩短分析周期并提供多元的可视化结果,避免人为分析的主观性。标准化分析模块覆盖多样本整合、细胞类型注释、肿瘤异质性、基因集发现与富集、细胞间相互作用、转录因子调控等个性化与高级分析点,上游衔接新格元CeleSCOPE™或其他软件处理下机数据结果,满足从下机数据到分析结果下载的闭环分析需求,支持多种单细胞平台数据。通过自动化注释软件和SynEcoSy™数据库的结合保证注释结果准确,并保留由新格元医学信息团队进行严谨的人工审核,在实现标准化避免人工误差的同时不牺牲任何准确性。CeleLens真正实现对单细胞数据分析的标准化和全流程的覆盖,为研究者们破解单细胞数据分析技术壁垒造成的应用阻力。
CeleLens Module 01:多样本整合分群与细胞亚型功能刻画
Module 01覆盖单细胞分析流程的重点难点:多样本整合分群与细胞质控、细胞注释和亚型功能刻画。这些流程涉及的分析工具数量众多,且每个工具都需要足够的经验和专业水平来调试出最佳参数进行分析,保障分析结果的质量。Module 01免去研究者对单个工具调试优化的过程,配备新格元经过多次研发测试调试出的最优参数,快速输出准确丰富的分析结果。
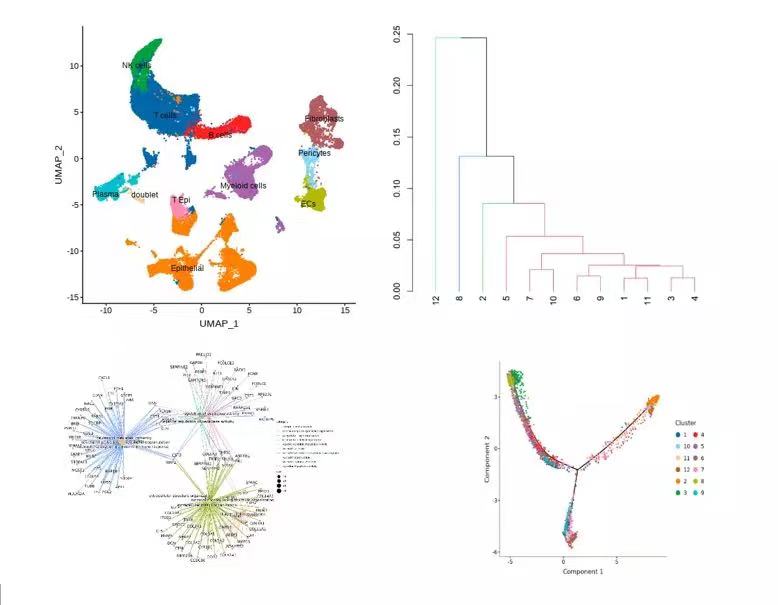
图注:左上,展示带有细胞类型标签的UMAP降维图;右上,展示亚型细分时各亚型的聚类树图,帮助更好的划分亚型;左下,根据功能富集分析结果绘制的通路网络图;右下,根据拟时序分析结果绘制各亚型分化关系图。
1)多样本整合分群和细胞类型注释
Module 01采用主流单细胞分析软件Seurat[1]进行多样本整合,同时也配备能够处理百万细胞数据级别的Scanpy[2]。二者都配备用项目经验优化过的参数对数据进行严格质控,得到高质量细胞进行降维分群和可视化展示。
分群后的细胞通过Cell-ID机器学习算法[3]进行自动注释,取代传统的人工注释,结果可靠且显著提速。基于新格元自有SynEcoSys™单细胞cell marker数据库,Cell-ID自动化注释目前可应用于人和小鼠两个物种,并覆盖广泛的器官、组织类型,能识别出非常全面的细胞类型,包括proliferating等状态的细胞。另一方面,Cell-ID还可选择已训练的的不同器官reference来进行自动化注释,能准确高效地注释出器官特异性的实质细胞以及基质和免疫细胞。自动注释结果经严谨的人工审核无误后可进入高级分析流程,逐步揭示样本内部的故事。
2)细胞亚型细分和功能评估
对初步分群后的细胞进一步细分亚型,有助于更细致地揭示样本中的异质性。细分出的亚型可通过基因表达的相似性构建聚类树(Hcluster Tree),进一步明确亚型划分,帮助发现稀有亚型。亚型功能刻画通过GO、KEGG和GSVA富集分析,以及UCell基因集打分方法实现[4,5,6]。细胞亚型功能富集分析结果由bar plot、dot plot、lolly plot、net plot、heat plot、map plot、UMAP plot等丰富的可视化方式呈现。
3)细胞亚型发育轨迹分析
发育轨迹分析构建细胞亚型之间随时间的相互转化轨迹,体现发育分化过程,可识别出细胞分化进程中的中间态或过渡态基因。新格元选用Monocle和CytoTRACE软件进行发育轨迹分析[7,8]。Monocle通过细胞关键基因的差异寻找细胞发育特征,构建随时间分布的分化谱系关系。CytoTRACE则通过转录的多样性评估单个细胞的分化潜能,根据分化潜能建立分化轨迹并明确分化方向。后续还可通过GeneSwitches、tradeSeq等工具挖掘不同轨迹分支的驱动基因。多样的可视化结果包括:pseudotime plot、Gene-cluster plot、Cell-cluster plot、genes plot、state plot、samples plot、density plot。
4)细胞亚型转录调控网络分析
新格元采用pySCENIC[9]进行转录调控网络分析。分析首先使用GRNBoost2基于基因的共表达,建立潜在的转录因子和靶基因调控关系,接下来使用cisTarget筛选存在直接靶向关系的潜在调节子,最后使用AUCell计算调节子的活性,并输出细胞类型或者样本特异性的调节子。
CeleLens Module 02:肿瘤异质性
Module 02主要进行肿瘤细胞标准化判定并结合肿瘤异质性分析。准确识别肿瘤细胞后,分析不同肿瘤来源或同一肿瘤中亚克隆细胞的遗传和表型特征多样性,包括基因表达的改变等,解析样本的瘤内异质性以及不同样本间的瘤间异质性。

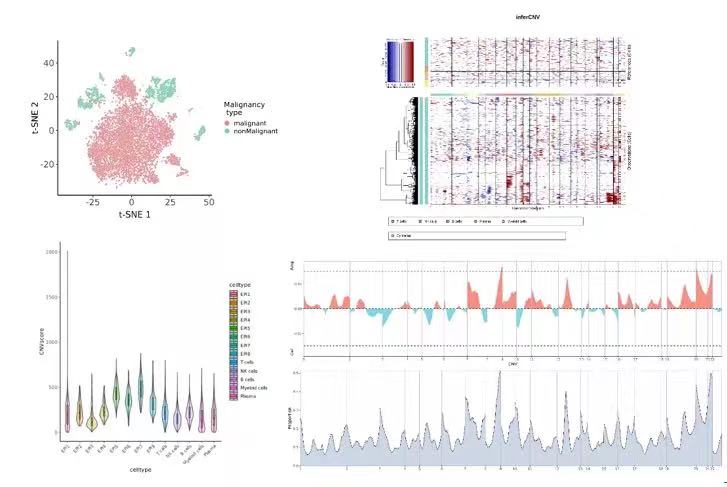
图注:左上,展示scCancer鉴定肿瘤细胞的结果;右上,肿瘤细胞inferCNV热图;左下,肿瘤细胞亚型CNVscore箱线图;右下,CNV特异性区段展示。
相较于正常细胞来说,一些癌种的肿瘤细胞缺乏与正常细胞区分的的完整marker体系,人工分析时难以精确判定肿瘤细胞。新格元通过优化并结合scCancer和copyKat软件来自动判定肿瘤细胞[10],提升准确性。对确定的肿瘤细胞进行inferCNV分析[11],展示染色体发生大规模插入和缺失的情况,通过heat plot、violin plot、box plot等多种可视化方法展示,可比较不同细胞类型中的CNV变异情况,或比较得出恶性程度更高的肿瘤亚型。
CeleLens Module 03:细胞间相互作用
细胞间相互作用是生长发育和疾病发生研究中的必要研究对象。Module 03进行细胞间相互作用的标准化分析,免去调试参考数据库与分析软件的过程,直接得到清晰的可视化结果。
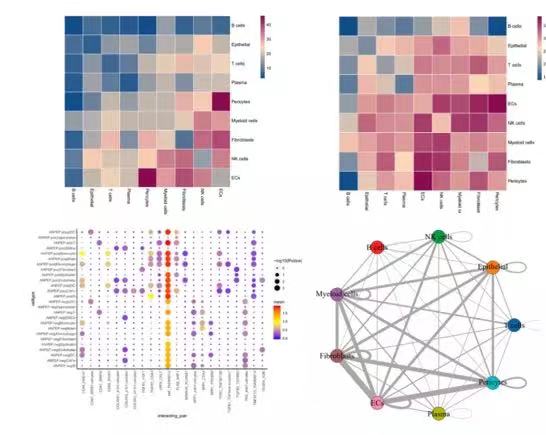
图注:左上,细胞间互作对数目热图;右上,细胞间互作对数目log转化后的热图;左下,细胞类型间互作对表达情况及显著性气泡图;右下,细胞间互作对数目网络图。
新格元细胞互作分析参考CellPhoneDB受配体作用数据库[12],可分析亚基层面的相互作用,包含以下三大类作用方式。得到清晰细致的细胞间相互作用关系,以heat plot、dot plot、net plot等进行展示。
· 直接接触(通过膜与膜结合的信号分子)
· 借助于信号分子(间接信息交流)
· 借助于特殊通道(相邻细胞之间),如胞间连丝
· 结合SynEcoSys™数据库中受配体信息展示少量感兴趣的受配体对,如细胞因子,免疫检查点等
CeleLens Module 04:基因集发现与富集
Module 04自动进行细胞特征基因集的发现,并对新发现或已知的基因集进行打分。每个基因集一般反应一种细胞特征/功能,如保障细胞基本生存,或恶性特征等等。然而目前已知的基因集只能反映很小一部分细胞功能,因此从转录中发现新的特征基因集能拓宽我们对基因在细胞中功能与协同作用的认识。在肿瘤细胞转录中拆解出恶性特征基因集能够辅助对恶性细胞和正常细胞的区分,或寻找恶性细胞的正常细胞来源。特征基因集发现是近几年发表的新兴方法,新格元比较调试后择优打包成module 04, 方便研究者将新型分析应用在工作中。
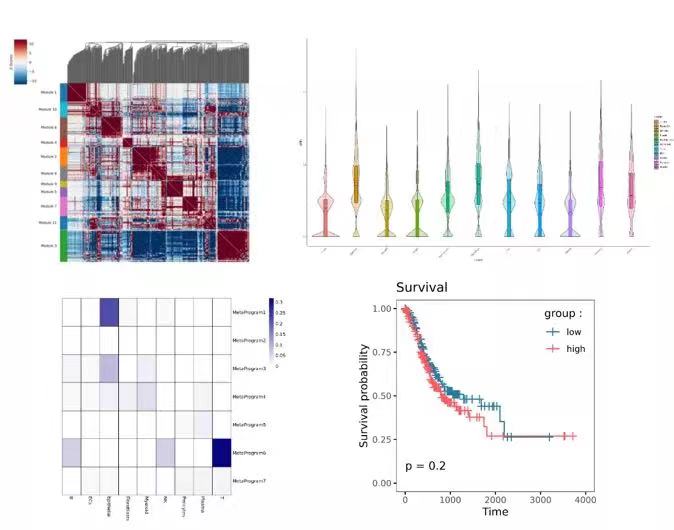
图注:左上,基因相关性热图;右上,各细胞指定基因集合Ucell得分小提琴图;左下,细胞类型与基因集合相似性热图,颜色深浅代表杰卡德相似性系数;右下,基因集评分生存曲线图。
新格元采用hotspot方法从数据中进行功能基因集(meta-program)发现[13]。由hotspot发现的基因集,或GO、KEGG、Reactome、MsigDB等公共数据集中的通路基因集、以及新格元团队从文献中收集的13+个特征基因集,可进行如下分析:
· UCell或AUC基因集打分,来展示每个基因集在细胞中的富集情况
· Jaccard相似性分析[14]展示不同基因集的相似程度,以及细胞类型间基因集活性的相似程度,通过细胞间的相似性可寻找肿瘤细胞潜在的的正常细胞起源
· 针对肿瘤样本,联合TCGA bulk RNA数据,进行基因集打分与生存预后和临床因素的关联分析,结果可对临床疾病的治疗进行指导,通过生存曲线图、不同样本分组中的差异分析箱线图、森林图及随临床因素改变的变化趋势图展示
工欲善其事,必先利其器。新格元CeleLens Module 01- 04 可依据分析需求自由搭配使用,用更短的时间输出更加丰富的结果。在单细胞数据分析操作简便化的同时,每个Module都采用新格元通过真实样本的经验不断优化得出的最佳软件、参考数据和参数搭配,保障分析结果精准,结果图表直接适配主流科研杂志常用的配色方案,提高科研工作效率。
参考文献
1. Stuart, T. et al. Comprehensive integration of single-cell data. Cell 177, 1888–1902 (2019).
2. Wolf, F. A., Angerer, P. & Theis, F. J. Scanpy: large-scale single-cell gene expression data analysis. Genome Biol. 19, 15 (2018)
3. Cortal, A., Martignetti, L., Six, E. et al. Gene signature extraction and cell identity recognition at the single-cell level with Cell-ID. Nat Biotechnol 39, 1095–1102 (2021).
4. Wu T, Hu E, Xu S, et al. clusterProfiler 4.0: A universal enrichment tool for interpreting omics data. Innovation (N Y). 2021;2(3):100141.
5. Hänzelmann S, Castelo R, Guinney J. GSVA: gene set variation analysis for microarray and RNA-seq data. BMC Bioinformatics. 2013;14:7.
6. Andreatta M, Carmona SJ. UCell: Robust and scalable single-cell gene signature scoring. Comput Struct Biotechnol J. 2021;19:3796-3798.
7. Xiaojie Qiu, Andrew Hill, Cole Trapnell et al (2017): Single-cell mRNA quantification and differential analysis with Census. Nature Methods
8. Gulati GS, Sikandar SS, Wesche DJ, et al. Single-cell transcriptional diversity is a hallmark of developmental potential. Science. 2020;367(6476):405-411.
9. Van de Sande B, Flerin C, Davie K, et al. A scalable SCENIC workflow for single-cell gene regulatory network analysis. Nat Protoc. 2020;15(7):2247-2276.
10. Guo W, Wang D, Wang S, Shan Y, Liu C, Gu J. scCancer: a package for automated processing of single-cell RNA-seq data in cancer. Brief Bioinform. 2021;22(3):bbaa127.
11. inferCNV of the Trinity CTAT Project. https://github.com/broadinstitute/inferCNV
12. Efremova M, Vento-Tormo M, Teichmann SA, Vento-Tormo R. CellPhoneDB: inferring cell-cell communication from combined expression of multi-subunit ligand-receptor complexes. Nat Protoc. 2020;15(4):1484-1506.
13. DeTomaso D, Yosef N. Hotspot identifies informative gene modules across modalities of single-cell genomics. Cell Syst. 2021;12(5):446-456.e9.
14. Dong R, Yang R, Zhan Y, et al. Single-Cell Characterization of Malignant Phenotypes and Developmental Trajectories of Adrenal Neuroblastoma. Cancer Cell. 2020;38(5):716-733.e6.








