产品介绍 | AccuraCode大规模转录组测序应用场景及其解决方法
发布时间:2022-11-30 14:43:09
一只蝌蚪变成青蛙,什么保持不变?什么发生了改变?
这是一个扣人心弦的问题,对它的回答,启发着我们对生命发育过程、疾病发生发展、药物研发筛选的理解。对它的回答,是一门学问也需要一个方法学框架,在这个框架内,转录组测序(RNA-seq)是一个正在被广泛应用的技术[1-4]。
Bulk RNA-seq中的差异表达基因筛选通常是最被关心的部分。然而常规的Bulk RNA-seq在大规模样品的差异表达基因的筛选上效率不高也存在批次偏差。所以基于常规RNA-seq实验方法,适用于大规模样品的转录组测序方法越来越多,例如BRB-seq[5]、Decode-seq[6]、Drug-seq[7]、AccuraCode®[8-9]和Prime-seq[10]。这类方法的核心思想是给每个样品的mRNA加上特定的barcode,然后混合样品建库,减去多个文库构建的需求,进而缩短周期和降低成本,减少批次效应。
AccuraCode®简介
AccuraCode®和Prime-seq的实验原理大致相同,是目前已知高通量转录组测序的最优实验方案[10]。二者均是基于scRNA-seq方法优化设计的,利用poly(A)富集,barcode和UMI标记生成3'标记的RNA-seq。自上市以来,AccuraCode®测序技术已被成功用于多个药效评估项目中,同时Prime-seq方法也已在17种生物中进行了验证并发表了22篇文章[8],足以说明该实验技术的稳定性。

Fig1. AccuraCode®实验方案
AccuraCode®采取的是一步法逆转录扩增反应(OneStep RT-PCR),高效获取各孔中cDNA分子。采取细胞系进行研究比较,从下面的结果可以看出,一步法的检出基因数与传统两步法相当。在保证了一致性的前提下,一步法的操作和时间成本更低。
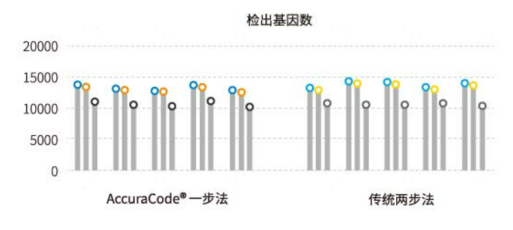
Fig2. 一步法逆转录扩增反应相对两步法的检出基因数比较
对于这些改进的RNA-seq高通量测序方法,大家最关心的问题是该实验方法得到的结果是否能与TruSeq保持高度的一致性,因为TruSeq已经广泛用于RNA-seq研究中。 通过以下与TruSeq比较中可以看出,无论是在read的mapping情况(Fig3. A)、基因检出数(Fig3. B),还是与Spike-in内参表达量的比较中(Fig3. C),两者均保持高度一致[1]。
此外,对于大多数RNA-seq实验,科研人员最关心的问题之一是寻找差异表达基因。通过比较两者的统计能力,在给定10m reads和FDR小于5%的情况下,两者的检测能力保持高度一致,并且会随着每组样品个数的提升而提升[10]。值得注意的是AccuraCode®和Prime-seq都使用了UMI进行表达定量,可以通过删除PCR扩增重复来减小技术重复差异[11]。
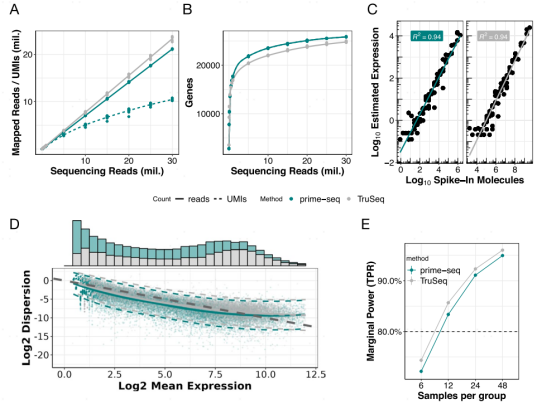
Fig3. 3端标记的RNA-seq和全长RNA-seq测序数据比较
AccuraCode®应用场景
AccuraCode®和Prime-seq可以应用于大规模的转录组定量测序(bulk RNA-seq),例如我们之前介绍的高通量药筛[9]和最近的研究热点eQTL[12-13]等,下方表格梳理了三种典型的RNA-seq测序方法的技术特点和应用范围:
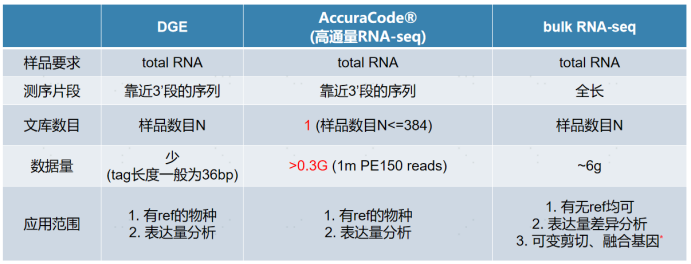
AccuraCode®于2021年上市[6],已成功应用于多个药企项目[14]。AccuraCode®的应用场景远不止于药筛,更能胜任其他关注RNA表达量的项目方案,让您获得更效率,更便宜,更少偏差的bulk RNA-seq解决方案。更多内容请关注:AccuraCode®高通量药物筛选RNA建库试剂盒. https://cn.singleronbio.com/product/detail-15.html
参考文献
1. Lister R, O’Malley RC, Tonti-Filippini J, Gregory BD, Berry CC, Millar AH, et al. Highly integrated single-base resolution maps of the epigenome in Arabidopsis. Cell. 2008;133:523–536.
2. Nagalakshmi U, Wang Z, Waern K, Shou C, Raha D, Gerstein M, et al. The transcriptional landscape of the yeast genome defined by RNA sequencing. Science. 2008;320:1344–1349.
3. Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nat Methods. 2008;5:621–628.
4. Holt RA, Jones SJ. The new paradigm of flow cell sequencing. Genome Res. 2008;18:839–846.
5. Alpern D, Gardeux V, Russeil J, et al. BRB-seq: ultra-affordable high-throughput transcriptomics enabled by bulk RNA barcoding and sequencing[J]. Genome biology, 2019, 20(1): 1-15.
6. Li Y, Yang H, Zhang H, et al. Decode-seq: a practical approach to improve differential gene expression analysis[J]. Genome biology, 2020, 21(1): 1-7.
7. Ye C, Ho D J, Neri M, et al. DRUG-seq for miniaturized high-throughput transcriptome profiling in drug discovery[J]. Nature communications, 2018, 9(1): 1-9.
8. 新品上市||AccuraCode®:新格元高通量药物筛选解决方案. https://mp.weixin.qq.com/s/qY-1eVxuk153UsgG9YR16g
9. AccuraCode®高通量药物筛选RNA建库试剂盒. https://cn.singleronbio.com/product/detail-15.html
10. Janjic A, Wange L E, Bagnoli J W, et al. Prime-seq, efficient and powerful bulk RNA sequencing[J]. Genome biology, 2022, 23(1): 1-27.
11. Fu Y, Wu P H, Beane T, et al. Elimination of PCR duplicates in RNA-seq and small RNA-seq using unique molecular identifiers[J]. Bmc Genomics, 2018, 19(1): 1-14.
12. Yazar, Seyhan, et al. "Single-cell eQTL mapping identifies cell type–specific genetic control of autoimmune disease." Science 376.6589 (2022): eabf3041.
13. Perez, Richard K., et al. "Single-cell RNA-seq reveals cell type–specific molecular and genetic associations to lupus." Science 376.6589 (2022): eabf1970.








