分析软件
sCircle®单细胞全长免疫受体数据分析
一、应声出笼
免疫组库(Immune Repertoire,IR)是指在指定时间,某个体循环系统中所有功能的多样性T细胞和B细胞的总和。在T细胞和B细胞中发现的受体分别被称为T细胞受体(T Cell Receptor,TCR)和B细胞受体(B Cell Receptor,BCR),细胞发育过程中存在复杂的V(D)J重排机制,导致TCR/BCR基因序列多样性极为丰富。 在单细胞水平同时检测转录组基因表达和免疫组库多样性,不但可以了解TCR和BCR克隆型,还可以联合转录组数据深入挖掘免疫机理,为免疫组学研究提供更高效的工具。
二、天工巧夺
新格元sCircle®单细胞全长免疫受体(TCR/BCR)建库试剂盒(sCircle®Single Cell Full Length Immuno-TCR/BCR Library Kit)基于Singleron单细胞平台,利用GEXSCOPE®微流控芯片捕获单细胞,通过反转录获得cDNA,cDNA分别用于转录组文库构建和全长免疫组库富集。
转录组文库构建 cDNA经片段化、连接接头等步骤后构建测序文库,获得3'端单细胞转录组文库。
全长免疫组库富集 首先将cDNA环化处理,将环化cDNA序列线性化,同时将序列中最为重要的V(D)J序列进行方向转化,富集之后获得全长V(D)J序列。
三、 聚焦优势
- 高通量、高分辨率、全流程——基于Singleron单细胞平台,实现海量单细胞水平上免疫组库多样性信息检测,可完成细胞分离、核酸捕获、环化、建库以及数据分析。
- 全长V(D)J捕获——可拼接获得免疫受体全长信息,对V(D)J基因序列进行组装和注释,获得单个免疫细胞克隆型配对序列信息,进而全面获取全长免疫组库多样性信息。
- 同时获得每个免疫细胞免疫受体全长信息和3’端单细胞转录谱信息。多元化应用场景,适配人源和鼠源免疫细胞。
- 可基于单细胞全长V(D)J序列构建系统发育树。
四、应用领域
- 肿瘤治疗-免疫微环境——免疫组库可揭示浸润T细胞的克隆多样性,发现起关键免疫作用的T细胞亚型,捕捉肿瘤发生时的免疫微环境变化,有利于寻找免疫治疗靶点,提高免疫治疗疗效。
- 抗体开发——利用免疫组库可以获得特征性的序列,大辐度缩短抗体开发的流程和时间。
- 自身免疫性疾病利用免疫组库技术可根据T细胞的克隆增殖情况,发现并分析异常表达的免疫细胞,寻找临床诊断标志物,为疾病的治疗提供依据。
- 移植和免疫重建——免疫组库技术可检测移植后T细胞库,根据T细胞的克隆增殖情况,发现起主要免疫作用的T细胞亚型,也可识别移植后感染或复发的高危人群。
- 用药及疫苗评估——免疫组库技术可在免疫治疗疗效的评估中,对用药前后的外周血样本进行评估,确认药物是否激发免疫反应及其药效情况。
五、sCircle单细胞全长V(D)J运行流程
1.全长免疫组库分析流程
新格元的全长免疫组库建库策略,通过一步环化和三轮富集,增加了 TCR 和 BCR 的比对率。那么在拿到全长免疫组库数据后,我们可以使用新版本 CeleScope1.11.0 内置的multi_flv_trust4 管线进行全长免疫组库数据分析。
2.管线流程
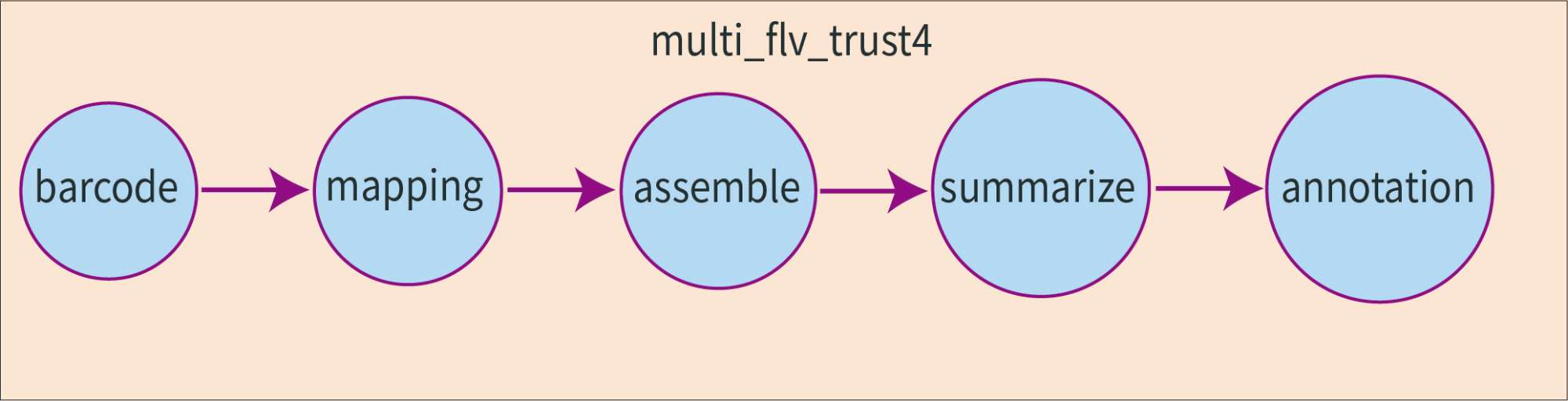
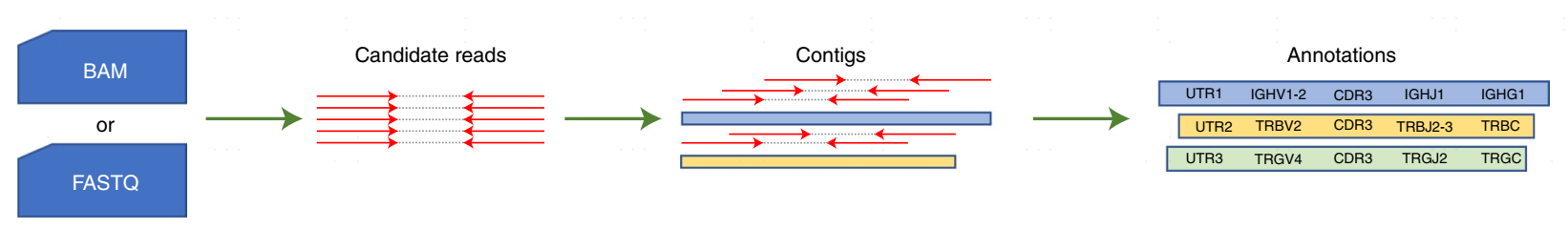
新格元将刘小乐教授开发的 Trust4 算法[1] 整合进了 CeleScope,它加快了传统免疫受体组装的速度,组装完成的细胞数目也有所增加。CeleScope内置了5个模块,模块对数据的处理顺序如上,接下来我们细述它们完成的工作。
-
Barcode: 步骤识别 reads 中的 barcode 和 UMI 并进行拆分;这里需要对免疫组库数据的 barcodes 进行反向填充以匹配 RNA 文库的 barcodes,最终仅保留匹配 RNA 文库中存在的 barcodes 对应的 reads。
-
Mapping: 步骤进行比对,提取候选 reads(Candidate reads) 进行组装
-
Assemble: 步骤完成从头组装,reads的重叠判定与重叠群更新的算法可以参考 Trust4 原文
-
Summarize: 完成对组装结果的过滤和Cell-calling(算法和转录组一致),这步是关键的质控环节并生成下游分析的常用文件(clonotypes.csv,),过滤可以分为三种类型:
-
基于 CDR3 的一般过滤:
滤掉非功能性 CDR3 (==在 cdr3 报告中显示 “out of frame”==) 或在核苷酸序列中包含 “N” 的 CDR3 序列;
确保 CDR3 的氨基酸序列以 C(Cys) 开头,序列长度大于等于5,同时确保 CDR3 氨基酸对应的核苷酸序列中没有终止密码子;
根据 UMI 阈值过滤低丰度 contigs。
-
DiffuseFrac filtering(需要提供 --diffuseFrac 选项)
如果细胞A的两条链的CDR3s和另一个细胞B的完全一致,且细胞A链丰度显著低于细胞B的,过滤A
-
目标细胞 barcode 过滤(需要提供参数 --target_cell_barcode)
细胞破裂等特殊情况会在非免疫细胞上组装出免疫受体,这种噪音会对 cell calling 造成干扰。如果提供参数 --target_cell_barcode,一个包含T/B细胞的barcodes文档(需要对RNA文库的细胞类型进行注释),我们会对目标细胞产生的contigs 获得的所有UMI counts乘以一个权重系数(默认是6)来从背景噪音中更好的识别信号
-
-
Annotation: 完成的工作分为两块,如果转录组匹配文库(match_dir)提供了 rds 和 auto-assign 信息,将会提供组装好的T/B细胞的图谱。另一个工作依靠 IMGT数据库(国际免疫遗传学数据库) 生成 VDJ 注释信息以及 html 中的克隆型表格和分布条形图。
#参考文献
[1]. Song, Li et al. “TRUST4: immune repertoire reconstruction from bulk and single-cell RNA-seq data.” Nature methods vol. 18,6 (2021): 627-630. doi:10.1038/s41592-021-01142-2
3.快速入门
CeleScope flv_trust4生成的单细胞免疫组库数据的基本流程,它执行细胞标签识别、提取候选reads序列、reads序列从头组装、组装结果的过滤及Cell-calling、V(D)J序列信息注释和克隆类型计数表格。
conda activate celescope
$ celescope flv_trust4 --help
usage: celescope flv_trust4 [-h] {sample,barcode,mapping,assemble,summarize,annotation} ...
Single-cell flv_trust4
positional arguments:
{sample,barcode,mapping,assemble,summarize,annotation}
optional arguments:
-h, --help show this help message and exit
$ celescope -v
1.11.0 # 版本号
- 下载测试数据与脚本
为了便于测试软件,我们在github上托管了测试数据(请注意,这些数据仅供测试用途,部分数据是人为生成的)。 celescope 所有 pipeline 的脚本模板和测试数据可以通过以下两条指令下载:
mkdir test_dir
cd test_dir
git clone https://github.com/singleron-RD/celescope_test_data.git
git clone https://github.com/singleron-RD/celescope_test_script.git
当然,也可以在gitee上下载:
mkdir test_dir
cd test_dir
git clone https://gitee.com/singleron-rd/celescope_test_data.git
git clone https://gitee.com/singleron-rd/celescope_test_script.git
所有的软件DEMO测试数据我们已经在celescoperna的教程中下载过,这里可以看一下vdj数据的结构,让我们看一看下载的测试数据
$ tree
.
├── fake_match_dir
│ └── 05.count
│ └── filtered_feature_bc_matrix
│ └── barcodes.tsv
├── fltest_R1.fq.gz
└── fltest_R2.fq.gz
- 下面是测试脚本:
cd flv_trust4
$ tree -L 2
.
|-- run_shell.sh
`-- flv_trust4.mapfile
六、单细胞免疫组库实操过程
单细胞免疫组库研究在实验过程中会构建一个转录组文库和一个免疫受体富集文库,因此数据分析也就分为两个环节: (1) 单细胞转录组分析 (2) 单细胞免疫受体分析 本篇文章内只介绍单细胞免疫受体数据分析,而celescope分析单细胞转录组数据的教程已在前期中介绍过。此外,如果只做免疫受体数据分析,可以单独建单细胞免疫组库进行分析,后续的分析流程是一致的。 在分析之前我们先要激活我们celescope软件的运行环境,可以使用conda activate celescope命令进行激活。
(base) singleron 13:20:48 ../../flv_trust4
$ conda activate celescope
(celescope) singleron 13:21:18 ../../flv_trust4
然后,接下来就是要使用的两个重要的配置文件,flv_trust4.mapfile和run_shell.sh
$ tree -L 1
.
|-- run_shell.sh
`-- flv_trust4.mapfile
1、用 multi_vdj 构建 celescope vdj 分析的 shell 脚本
- 配置 mapfile文件--mapfile 是multi_vdj下的参数,需要提供一个制表符分隔 (tab-delimited) 的文本文件。mapfile 的每一行代表双端 (paired-end) fastq文件。
# 这是我们写好的全长免疫组库的mapfile
fltest PATH/TO/YOUR/SEQENCING/DIRECTORY sample_name PATH/TO/MATCHED/DIRECTORY
- mapfile说明
第一列:mapfile 是制表符分隔的四列式文本文件,第一列是测序下机数据的前缀,以下是关于前缀的示例:
# sample1 有 2 个双端测序 fastq 文件位于两个不同的目录下(fastq_dir1 和 fastq_dir2),前缀分别为 fastq_prefix1 和 fastq_prefix2;
# sample2 有 1 个双端测序 fastq 文件位于 fastq_dir1,前缀为 fastq_prefix3
$cat ./../my.mapfile
fastq_prefix1 fastq_dir1 sample1
fastq_prefix2 fastq_dir2 sample1
fastq_prefix3 fastq_dir1 sample2
$ls fastq_dir1
fastq_prefix1_1.fq.gz fastq_prefix1_2.fq.gz
fastq_prefix3_1.fq.gz fastq_prefix3_2.fq.gz
$ls fastq_dir2
fastq_prefix2_1.fq.gz fastq_prefix2_2.fq.gz
第二列: 第一列 Fastq 文件所在目录的路径
第三列: 样本名称(自拟),将作为所有输出文件的前缀
第四列: 第四列在不同的实验类型中意义不同。全长免疫组库需要的是对应转录组pipeline生成的结果文件夹路径 温馨提示一下:
multi_flv_trust4 不能同时完成转录组和免疫组库的拆库定量,转录组需要单独建库并使用 multi_rna 获得转录组表达矩阵。在使用 multi_rna 获得全长免疫组库的 matched_dir 时,需要指定 --chemistry 为 flv_rna。
- 另一个是 shell 脚本文件:run_shell.sh
# multi_flv_trust4 配置
multi_flv_trust4 \
--mapfile ./test.mapfile \
--ref hg38 \
--thread 8 \
--seqtype BCR \
2、生成命令脚本
(1)运行刚编辑好的shell脚本run_shell.sh
$ sh run_shell.sh
(2)运行完以后就可以自动生成一个名称为shell的文件目录。
$ tree -L 1
.
|-- run_shell.sh
|-- shell
`-- flv_trust4.mapfile
shell文件夹中会有一个以flv_trust4_test命名的脚本运行存储数据的目录,以及一个运行的shell脚本flv_trust4_test.sh,flv_trust4_test.sh脚本中的每行指令对应每一步分析(质控报告的每一部分数据)。
- 参数说明:
以上为必须参数,--ref是我们组装contigs时需要的参考数据集,被我们打包在 CeleScope 中,您也可以在GitHub上看下我们提供了哪些数据集:
CeleScope/celescope/tools/trust4/database at master · singleron-RD/CeleScope (github.com)
- --seqtype 有 TCR 和 BCR 两种
3、投递shell脚本
进入到shell目录中,就可以运行脚本flv_trust4_test.sh,然后在终端命令行中输入flv_trust4_test.sh。那么程序就会在当前的终端界面运行。但是,如果在当前的终端界面中进行运行,终端界面就不能关闭,也不能掉线。那么,为了避免这种情况的发生,我们可以使用nohup将运行脚本提交到后台运行,执行nohup sh flv_trust4_test.sh &,然后会生成一个nohup.out的日志文件,该文件将会记录该流程上的基本执行过程。
$ tree -L 1
.
|-- flv_trust4_test
|-- flv_trust4_test.sh
`-- nohup.out
如果对每一步做了什么感兴趣,可以单独运行查看,cat vdj_test.sh 里面是:
# multi_flv_trust4生成的分析脚本
celescope flv_trust4 sample --outdir .//flvtest/00.sample --sample flvtest --thread 10 --chemistry auto --fq1 /mnt/sdd/workstation/celescope_test/celescope_test_data/flv/fltest_R1.fq.gz
celescope flv_trust4 barcode --outdir .//flvtest/01.barcode --sample flvtest --thread 10 --chemistry auto --lowNum 2 --allowNoLinker --fq1 /mnt/sdd/workstation/celescope_test/celescope_test_data/flv/fltest_R1.fq.gz --fq2 /mnt/sdd/workstation/celescope_test/celescope_test_data/flv/fltest_R2.fq.gz --match_dir /mnt/sdd/workstation/celescope_test/celescope_test_data/flv/fake_match_dir
celescope flv_trust4 mapping --outdir .//flvtest/02.mapping --sample flvtest --thread 10 --ref hg38 --seqtype TCR --barcodeRange "0 23 +" --umiRange "24 -1 +" --match_fq1 .//flvtest/01.barcode/flvtest_1.fq --match_fq2 .//flvtest/01.barcode/flvtest_2.fq
celescope flv_trust4 assemble --outdir .//flvtest/03.assemble --sample flvtest --thread 10 --ref hg38 --seqtype TCR --barcodeRange "0 23 +" --umiRange "24 -1 +" --candidate_fq .//flvtest/02.mapping/flvtest_bcrtcr.fq
celescope flv_trust4 summarize --outdir .//flvtest/04.summarize --sample flvtest --thread 10 --seqtype TCR --coef 5 --expected_target_cell_num 3000 --target_weight 6.0 --assemble_out .//flvtest/03.assemble/assemble --fq2 .//flvtest/01.barcode/flvtest_2.fq --match_dir /mnt/sdd/workstation/celescope_test/celescope_test_data/flv/fake_match_dir
celescope flv_trust4 annotation --outdir .//flvtest/05.annotation --sample flvtest --thread 10 --seqtype TCR --summarize_out .//flvtest/04.summarize --match_dir /mnt/sdd/workstation/celescope_test/celescope_test_data/flv/fake_match_dir
4、结果目录
运行完成之后,可以看一下结果文件,其目录如下:
.
├── 00.sample
│ └── stat.txt
├── 01.barcode
│ ├── flvtest_1.fq
│ ├── flvtest_2.fq
│ └── stat.txt
├── 02.mapping
│ ├── flvtest_bcrtcr_bc.fa
│ ├── flvtest_bcrtcr.fq
│ ├── flvtest_bcrtcr_umi.fa
│ ├── flvtest_TRA_bc.fa
│ ├── flvtest_TRA.fq
│ ├── flvtest_TRA_umi.fa
│ ├── flvtest_TRB_bc.fa
│ ├── flvtest_TRB.fq
│ ├── flvtest_TRB_umi.fa
│ └── stat.txt
├── 03.assemble
│ └── assemble
│ ├── count.txt
│ ├── flvtest_annotate.fa
│ ├── flvtest_assembled_reads.fa
│ ├── flvtest_assign.out
│ ├── flvtest_barcode_filter_report.tsv
│ ├── flvtest_barcode_report.tsv
│ ├── flvtest_cdr3.out
│ ├── flvtest_filter_report.tsv
│ ├── flvtest_report.tsv
│ └── temp
├── 04.summarize
│ ├── clonotypes.csv # 下游分析文件
│ ├── count.txt
│ ├── flvtest_all_contig.csv
│ ├── flvtest_all_contig.fasta
│ ├── flvtest_b.csv
│ ├── flvtest_filtered_contig.csv # 下游分析文件
│ ├── flvtest_filtered_contig.fasta # 下游分析文件
│ ├── flvtest_t.csv
│ └── stat.txt
├── 05.annotation
│ └── stat.txt
└── flvtest_report.html # html报告
8 directories, 34 files
当我们运行完以后,就可以得到一个单细胞免疫组库的网页版报告。
- 质控报告的样本和软件的基本信息
- 数据质控信息
- 一致性序列
- VDJ基因比对
- 生成克隆类型计数
- 进行UMI计数以及细胞数目评估和克隆型数据统计。
- 克隆类型详细信息统计 好啦,以上就是一个完整的新格元sCircle单细胞全长V(D)J免疫受体解决方案的生信分析教程,接下来就可以进行深入的全长免疫组库克隆型多样性分析啦!!!
本文仅做软件安装测试使用,更多软件更新信息参见:https://github.com/singleron-RD/CeleScope








